Ist die Evolution von Grippeviren vorhersehbar?
Richard Neher
Biozentrum, University of Basel
slides at neherlab.org/201803_SeniorenUni_de.html
Verschiedene Viren
 tobacco mosaic virus
(Thomas Splettstoesser, wikipedia)
tobacco mosaic virus
(Thomas Splettstoesser, wikipedia)
bacteria phage (adenosine, wikipedia)
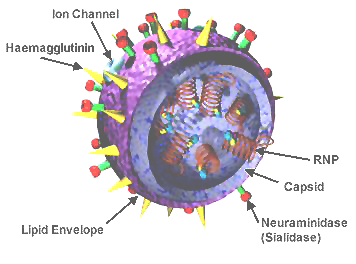
influenza virus wikipedia
human immunodeficiency virus (HIV) wikipedia
- Viren benötigen Wirtszellen zur Replikation
- sind wenig mehr als Genom + Kapsid
- ihr Genom ist 5-200Tsd Basen lang (+Ausnahmen)
- zahlenmäßig dominierenden Organismen $\sim 10^{31}$
Influenza virus
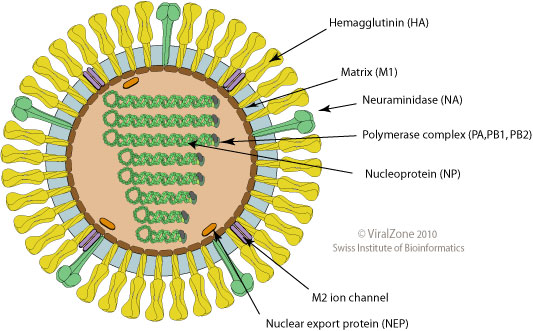
- Oberflächenproteine Hämagglutinin (HA) und Neuraminidase (NA)
- Influenza A Virus
- verbreitet in Vögeln und Säugetieren
- Viele Subtypen der Oberflächenproteine
- H3N2, H1N1, H7N9, H5N1
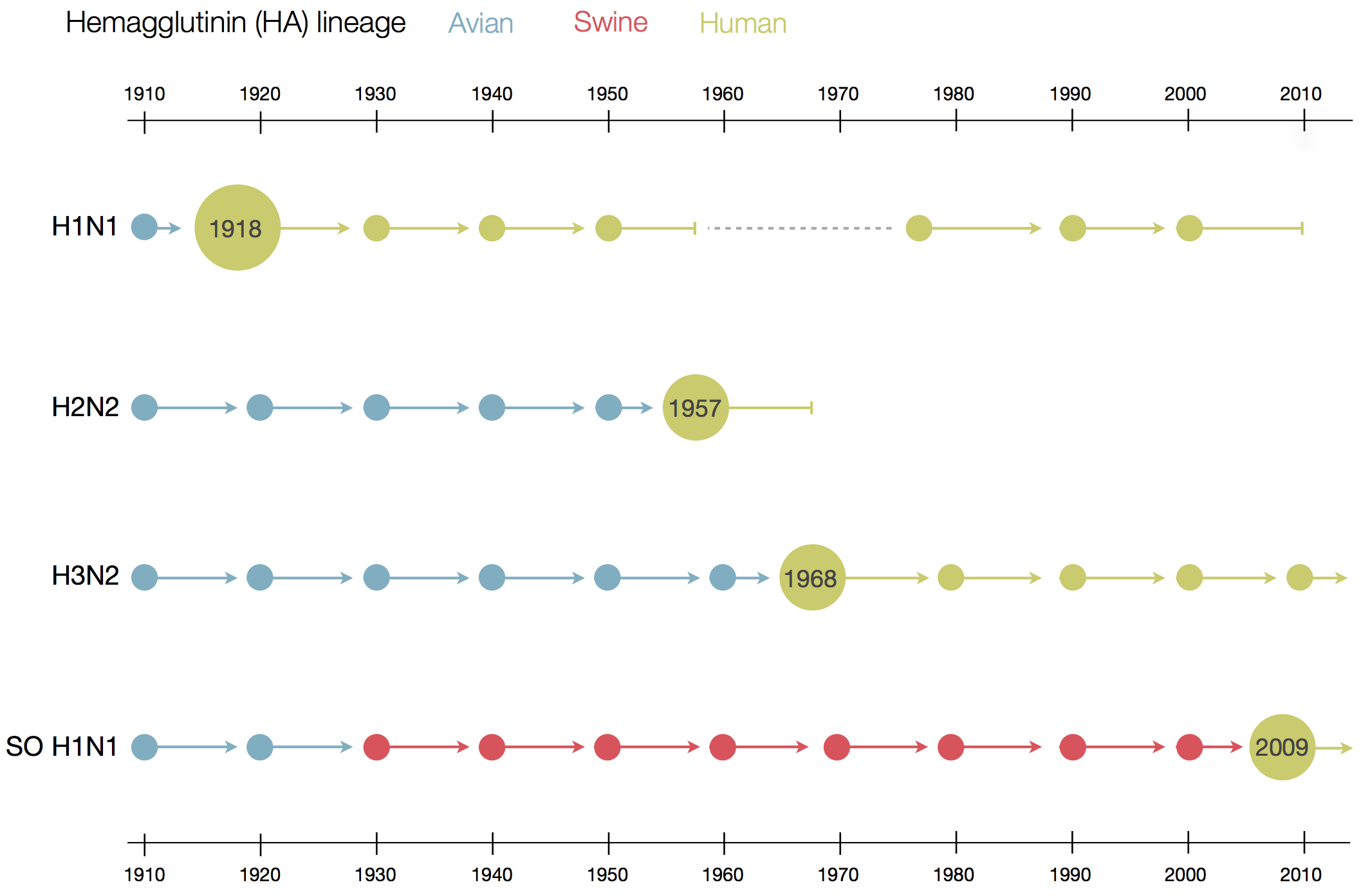
- H3N2, H1N1 zirkulieren im Menschen
- Influenza B Virus
- infiziert fast nur Menschen
- zwei Linien die sich vor 30-40 Jahren geteilt haben
- B/Victoria und B/Yamagata
- nur eine Variante ist in der tri-valenten Impfung enthalten
Influenza A Viren des Menschen
Wöchentliche Grippe-Fallzahlen in den USA
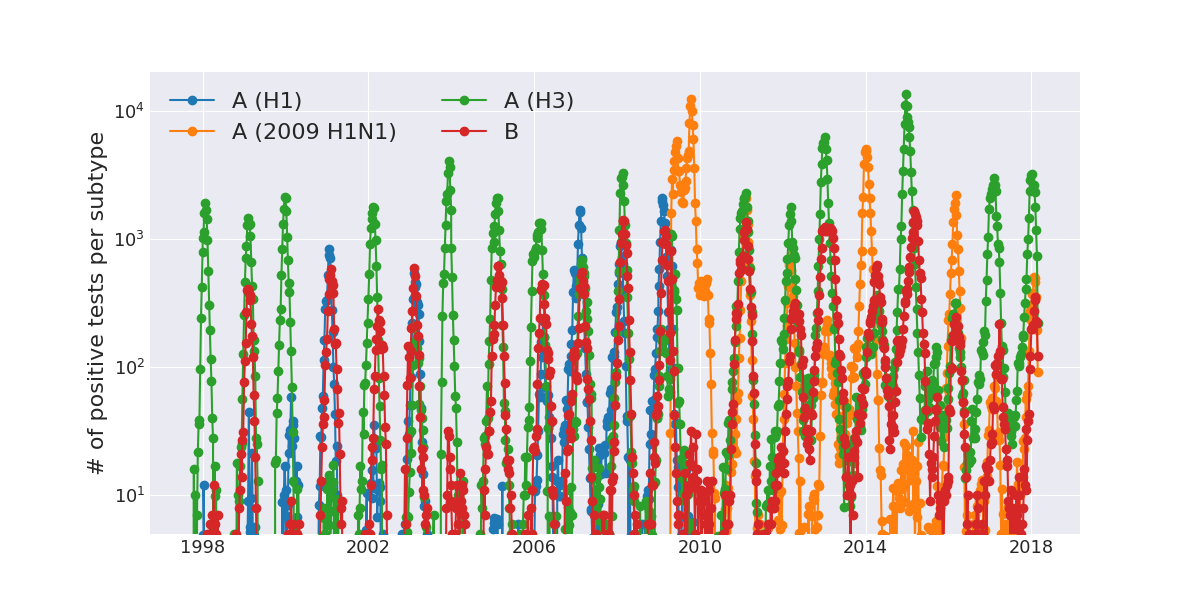 Data by the US CDC
Data by the US CDC
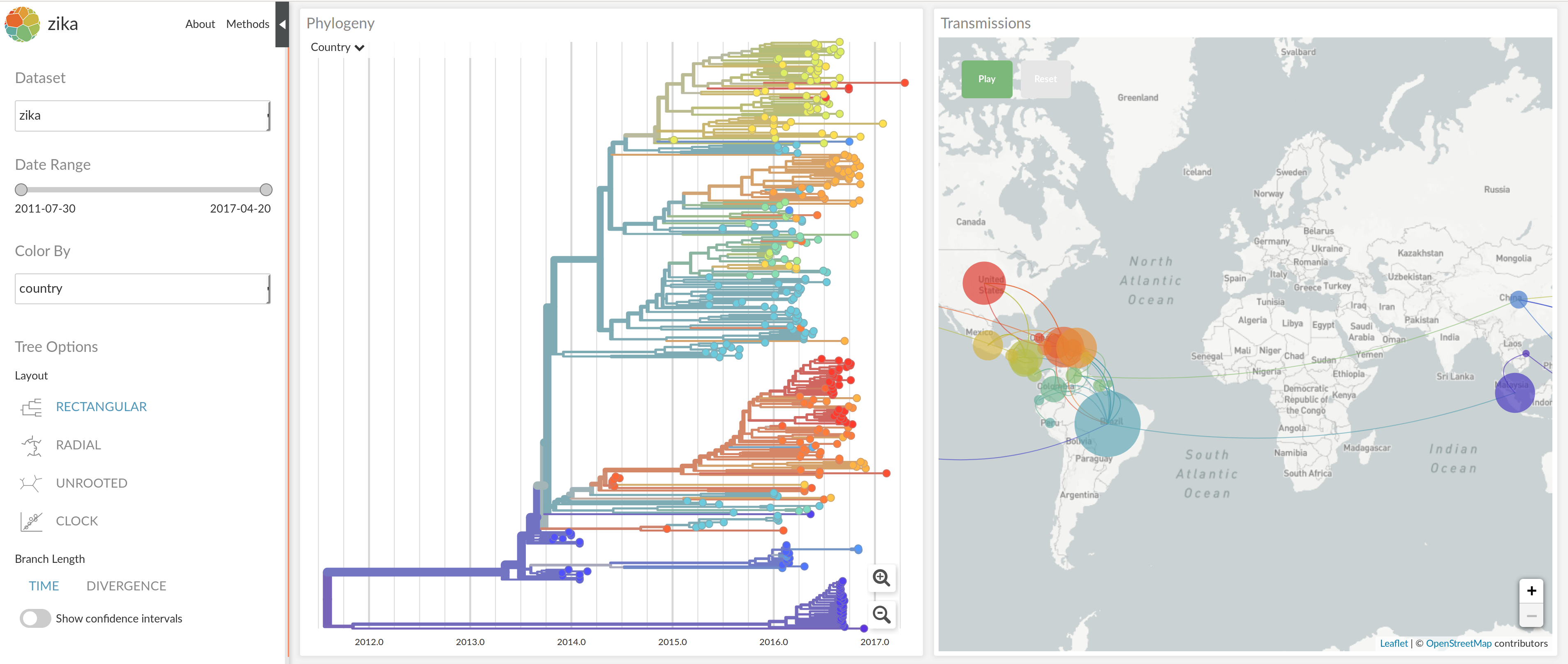
Genom-Sequenzen verraten die Ausbreitung und Evolution des Virus
A/Brisbane/100/2014
GGATAATTCTATTAACCATGAAGACTATCATTGCTTT...
A/Brisbane/1000/2015
GGATAATTCTATTAACCATGAAGACTATTATTGCTTT...
A/Brisbane/1/2017
GGATAATTCTATTAACCATGAAGACTATCATTGCTTT...
... Tausende Sequenzen mehr...
GGATAATTCTATTAACCATGAAGACTATCATTGCTTT...
A/Brisbane/1000/2015
GGATAATTCTATTAACCATGAAGACTATTATTGCTTT...
A/Brisbane/1/2017
GGATAATTCTATTAACCATGAAGACTATCATTGCTTT...
... Tausende Sequenzen mehr...
Phylogenetische Analyse von Virus-Genomen
RNA Viren haben hohe Mutationsraten. Ein Genom mutiert all paar Wochen.
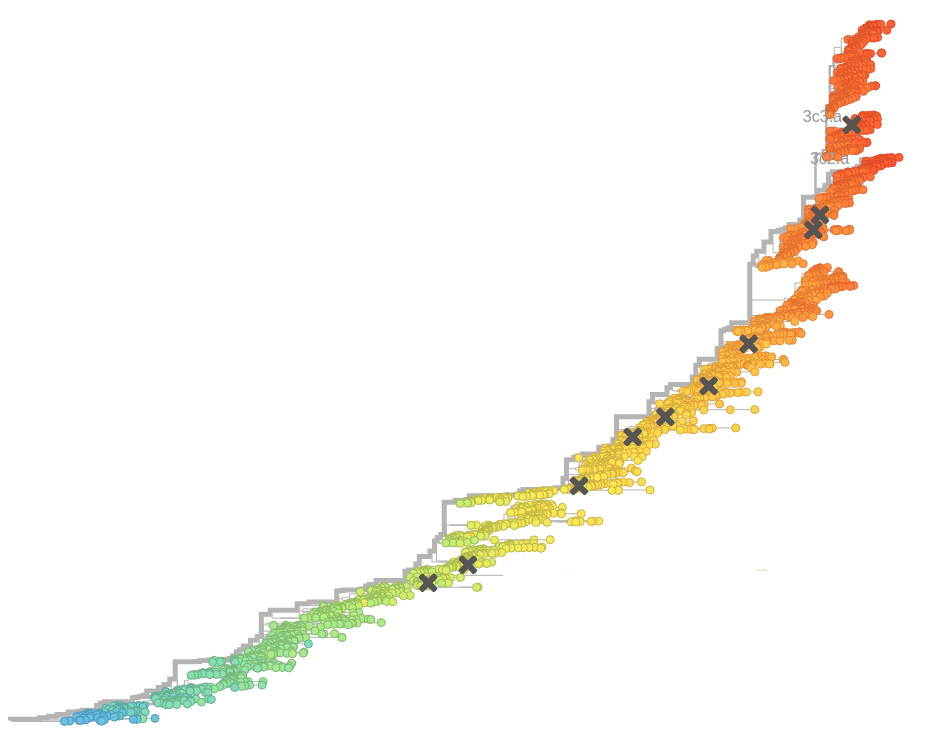
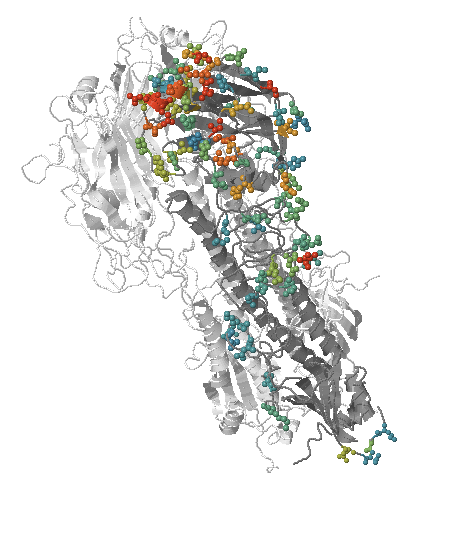
- Influenza Viren verändern sich und weichen so dem Immunsystem aus.
- Impfstoffe müssen laufend aktualisiert werden
nextflu.org
joint work with Trevor Bedford & his lab
Ist Evolution von Grippe vorhersehbar?
- Extrapolation
- Vorhersagen durch Vergleich mit historischen Mustern
- Erfolgreiche Varianten anhand des Verzweigungsmuster detektieren
- Antigenische Eigenschaften zirkulierender Viren
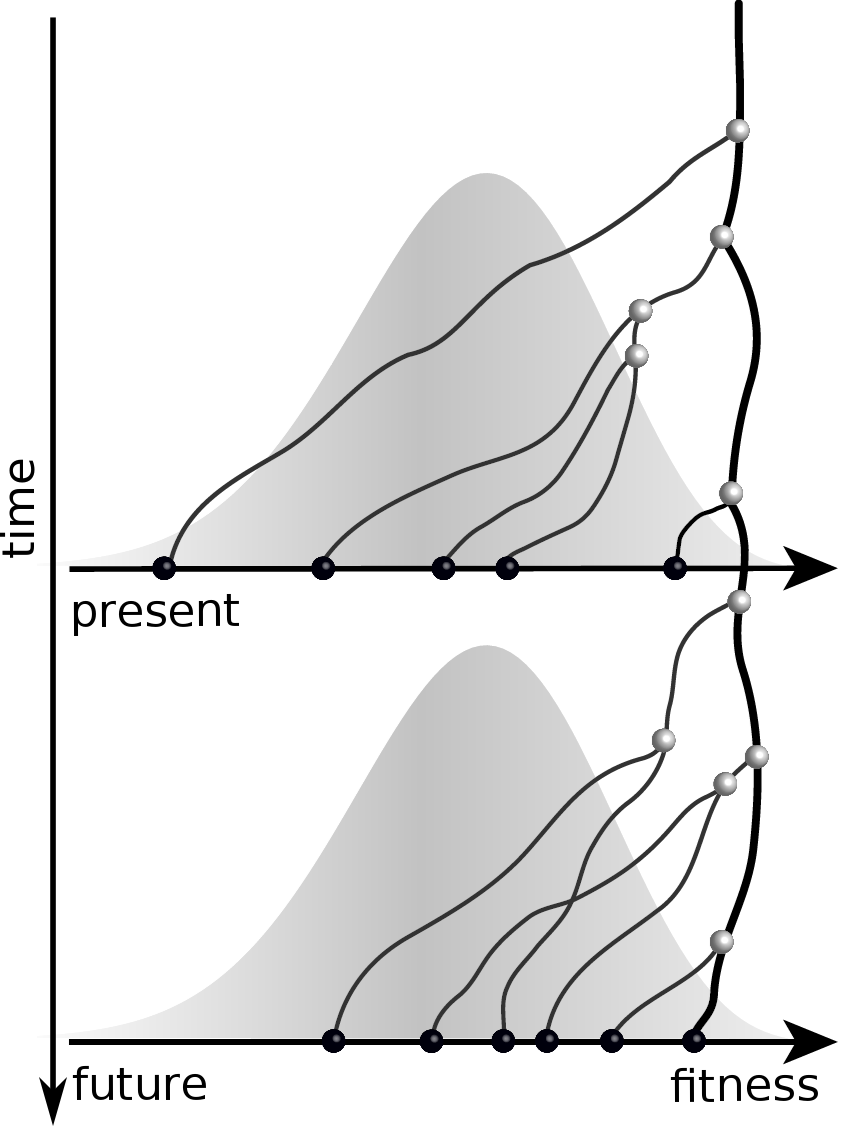
Fitness Inferenz anhand des Baums
$$P(\mathbf{x}|T) = \frac{1}{Z(T)} p_0(x_0) \prod_{i=0}^{n_{int}} g(x_{i_1}, t_{i_1}| x_i, t_i)g(x_{i_2}, t_{i_2}| x_i, t_i)$$
RN, Russell, Shraiman, eLife, 2014
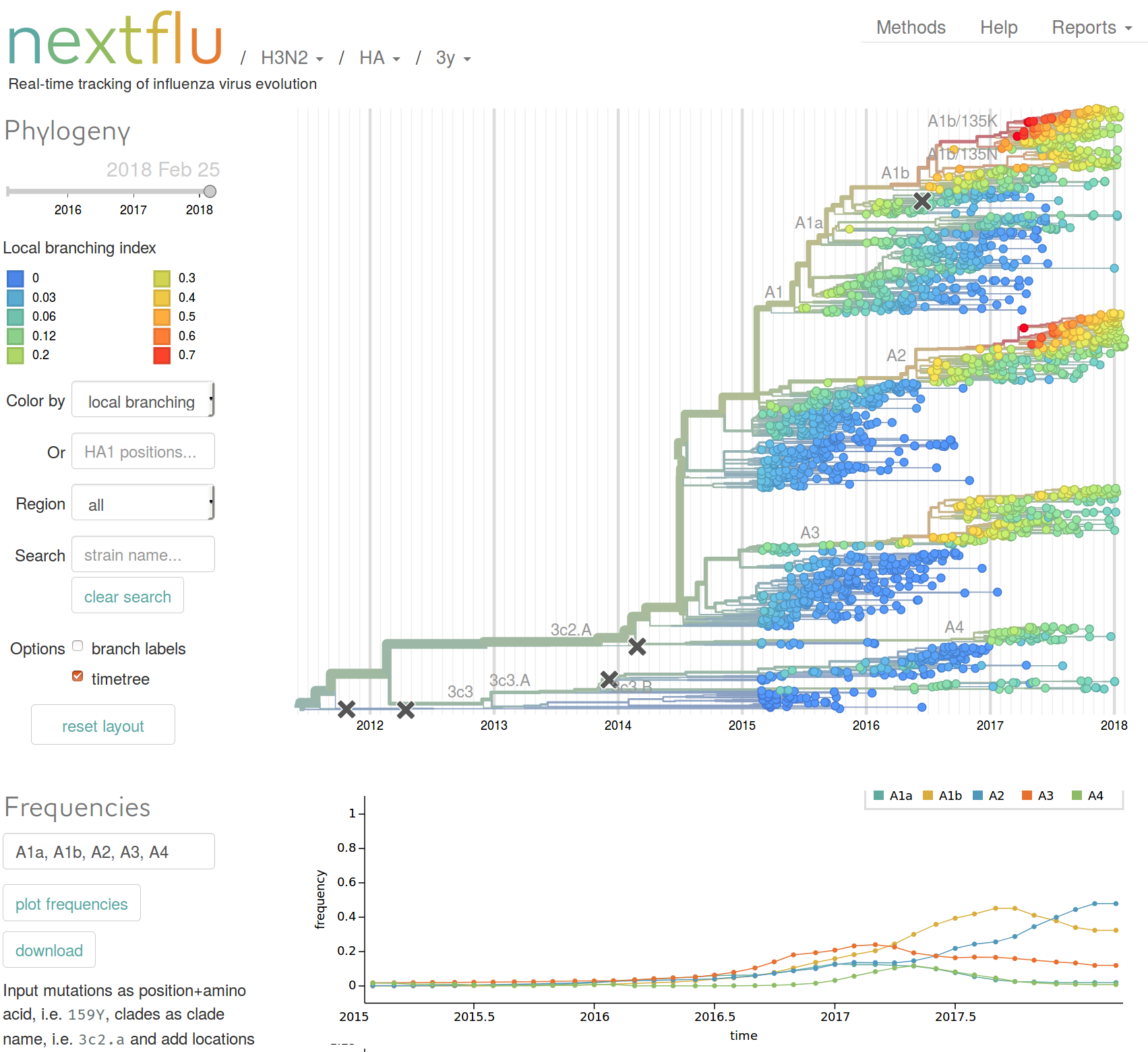
Vorhersage der dominierenden H3N2 Influenza Variante
- Nicht Grippe-spezifisch
- Wie kann das Model weiter verbessert werden? (siehe z.B. das Model von Luksza & Laessig)
- Anwendbarkeit in anderen Zusammenhängen?
nextstrain.org
joint work with Trevor Bedford & his lab
Zusammenfassung
- RNA Viruses evolvieren schnell
- Das Virusgenom verändert sich alle paar Wochen
- Aus Genomsequenzen kann Dynamik und Ausbreitung des Virus rekonstruiert werden
- Algorithm können vorhersagen, welche Grippe-Varianten nächste Saison dominiert
- Automatische real-time Analyse kann helfen Ausbrüche einzudämmen
Gemeinsame Arbeit mit....



- Boris Shraiman
- Colin Russell
- Trevor Bedford



nextstrain.org






- Trevor Bedford
- Colin Megill
- Pavel Sagulenko
- Sidney Bell
- James Hadfield
- Wei Ding



